中国农科院生物所和新加坡国立大学合作团队揭示拟南芥全基因组6mA修饰图谱
提示:还没关注?点上方「BioArt植物」↑
近年来,围绕DNA甲基化修饰调控生物发育和基因表达的研究是表观遗传的热点,5mC的产生机制和生物学功能已有较多研究,然而DNA上的另一种修饰6mA(6-甲基腺嘌呤)长期以来进展较少。近日,中国农业科学院生物技术研究所谷晓峰研究组和新加坡国立大学生物系及淡马锡生命科学研究所俞皓研究组合作在Developmental Cell杂志发表了题为“DNA N6-Adenine Methylation in Arabidopsis thaliana”的研究论文。该研究利用PacBio三代测序技术获得了模式植物拟南芥的全基因组6mA修饰图谱,解析了其分布模式和潜在功能。该成果将为进一步研究DNA 5mC和6mA甲基化协同调控机制及应用提供依据。

DNA甲基化是表观遗传研究领域的一个重要方面,胞嘧啶甲基化(5-methylcytosine,5mC)是研究最为深入的一种,在调控基因表达、发育、响应环境信号等多个方面起着关键作用,近年来逐渐揭示了DNA 第2 种修饰腺嘌呤甲基化(N6-methyldeoxyadenosine, 6mA)的重要作用【2-7】。从2015年《Cell》上同期3篇论文报道6mA在果蝇、线虫和衣藻的全基因组分布模式和功能开始,逐渐揭示其在真核生物中的功能,是表观遗传研究一个新热点领域【5-7】。
该研究利用Dot-Blot和LC-MS/MS等方法研究表明6mA 存在于拟南芥基因组DNA上,且在拟南芥不同发育时期和不同组织中是动态变化的。进一步利用三代单分子实时(Single-Molecule Real-Time,SMRT)测序方法绘制了拟南芥不同发育时期的全基因组6mA 单碱基分布图谱,SMRT的结果得到了LC-MS/MS和6mA-IP-qPCR的验证,并且发现在越靠近着丝粒区域表现出越高的6mA丰度和轻微降低的平均甲基化水平(图1)。
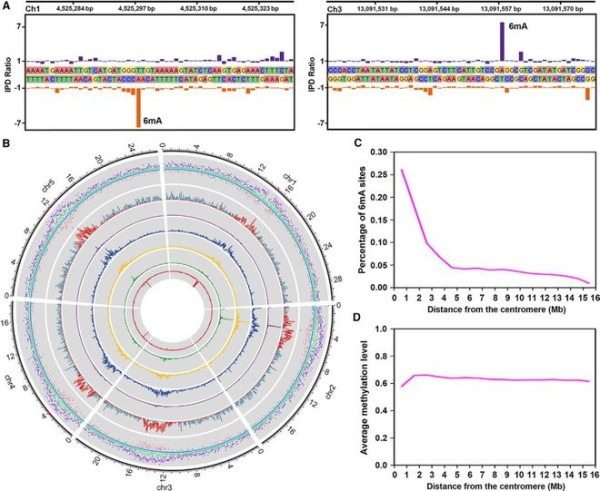
图1 DNA 6mA的全基因组分布
通过分析6mA在全基因组内不同的区域(5’UTR、Exon、Intron、3’UTR区)和修饰基因的不同类型(Protein coding、miRNA、snoRNA等),结果显示6mA在基因body区域分布更丰富,主要富集在exon区域,大多数6mA修饰的基因是蛋白编码基因。并进一步鉴定了植物特异和真核生物相对保守的motif。将6mA修饰基因与RNA-seq得到的基因表达的联合分析,结果表明6mA与拟南芥中活跃表达的基因相关联,其调控基因分布重要的生物学途径,可能参与植物种子形成、营养生长、光合作用、逆境适应等生物学功能。
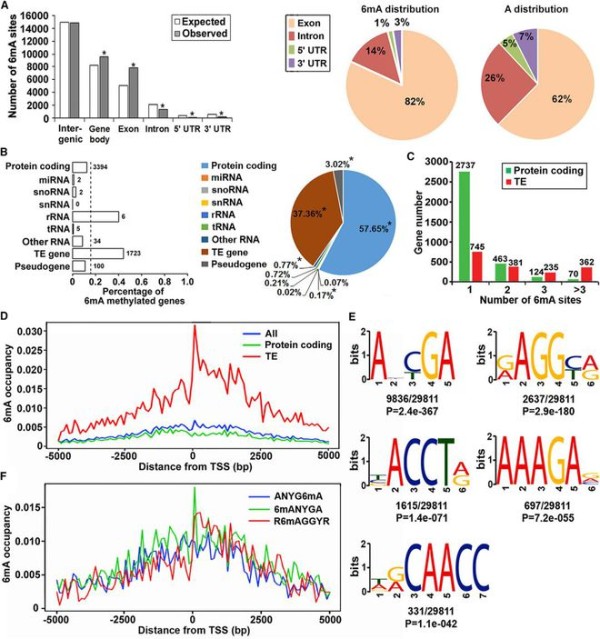
图2 DNA 6mA 基因分布特征
该研究首次揭示了植物DNA 6mA甲基化的全基因组分布和生物学功能,具有重要的科学意义,为系统解析DNA甲基化调控植物发育和环境适应性的机制开拓了新领域。
参考文献:
1. Liang Zhe 1, Shen Lisha 1, Cui Xuean1, Bao Shijie, Geng Yuke, Yu Guoliang, Liang Fang, Lu Tiegang, Gu Xiaofeng* and Yu Hao*. DNA N6-adenine methylation in Arabidopsis thaliana. Developmental Cell, 2018, 45:1-11
2. Mondo SJ, Dannebaum RO, Kuo RC, Louie KB, Bewick AJ, LaButti K, Haridas S, Kuo A, Salamov A, Ahrendt SR, Lau R, et al. Widespread adenine N6-methylation of active genes in fungi. Nat Genet, 2017, 49: 964-968.
3. Wu TP, Wang T, Seetin MG, Lai Y, Zhu S, Lin K, Liu Y, Byrum SD, Mackintosh SG, Zhong M, Tackett A, Wang G, et al. DNA methylation on N6-adenine in mammalian embryonic stem cells. Nature, 2016, 532: 329-333.
4. Liu J, Zhu Y, Luo GZ, Wang X, Yue Y, Wang X, Zong X, Chen K, Yin H, Fu Y, Han D, Wang Y, Chen D, He C. Abundant DNA 6mA methylation during early embryogenesis of zebrafish and pig. Nat Commun, 2016, 7: 13052.
5. Fu Y, Luo GZ, Chen K, Deng X, Yu M, Han D, Hao Z, Liu J, Lu X, Doré L, Weng X, Ji, Q, Mets L, He C. N6-Methyldexoyadenisine Marks Active Transcription Start Sites in Chlamydomonas. Cell, 2015, 161: 879-892.
6. Greer EL, Blanco MA, Gu L, Sendinc E, Liu J, Aristizábal-Corrales D, Hsu CH, Aravind L, He C, Shi Y. DNA methylation on N6-adeninein C. elegans. Cell, 2015, 161: 868-878.
7. Zhang G, Huang H, Liu D, Cheng Y, Liu X, Zhang W, Yin R, Zhang D, Zhang P, Liu J, Li C, Liu B, Luo Y, Zhu Y, Zhang N, He S, He C, Wang H, Chen D. N6-Methyladenine DNA modification in Drosophila. Cell, 2015, 161: 893-906.
点击底部“阅读原文”可查看论文链接。


网址:中国农科院生物所和新加坡国立大学合作团队揭示拟南芥全基因组6mA修饰图谱 https://m.mxgxt.com/news/view/1367974
相关内容
Nature Commun|南方科大宋毅团队揭示植物塑造抗旱微生物组的遗传和生态学机制祝贺!Nature:王红阳院士等团队揭示中国人群肝癌全基因组变异及演化特征
生物医学交叉研究院何新建实验室揭示拟南芥SAS复合体调控花发育的机制
中国农业科学院储明星研究员应邀为我院师生作学术报告
化学学院雷晓光团队与合作者破解抗癌明星天然药物紫杉醇的生物合成难题
中国科学院分子植物科学卓越创新中心王二涛研究组与合作者揭示根际微生物维持大豆产量的机制
动物科学学院学术报告(202310)
我国科学家即将绘制完成第一张黄种人基因组图谱
植物干细胞:两个明星基因的“星星”相“吸”—新闻—科学网
新研究填补全球基因组“南方盲区”

